【初心者向け】NCBIデータベースの概要と基本的な検索方法[バイオ/生命科学系DB解説②]

登録されたデータを誰もが無償で自由に利用できるデータベースとして、公的なデータベースが作られ公開されています。
米国では国立バイオテクノロジー情報センター(NCBI)、欧州では欧州バイオインフォマティクス研究所(EBI)、日本では国立遺伝学研究所DDBJセンターが中心となって整備し、維持されています。
今回はNCBIのデータベースの概要と検索方法の基本についてご紹介します。
1.NCBIとは?
NCBI(National Center for Biotechnology Information; 国立バイオテクノロジー情報センター)は、NLM(National Library of Medicine;アメリカ国立医学図書館)の一部門として、データベースの構築や運用を行う研究組織です。
塩基配列やアミノ酸配列を含む多数の生命科学・バイオインフォマティクス研究に用いられるデータベースが存在し、NCBIのサイトは生命科学・医学の様々な情報を検索するためのポータルサイトとして広く利用されています。
検索システムのEntrezによりNCBIのデータベースを統一的に検索することや、分野を選んで検索することができます。
(※NCBIのURL:https://www.ncbi.nlm.nih.gov/)
2.NCBIデータベースの概要
NCBIでは、以下の主要なものをを含め、多数のデータベースが提供されています。
- 核酸データベース: GenBank/Nucleotide/RefSeq
- ゲノムデータベース: Genome
- 多型データベース: dsSNP
- ヒトの遺伝子と疾患のデータベース: OMIM
- 遺伝子発現データベース: GEO
- タンパク質配列や構造のデータベース: Protein/Structure
- 文献データベース: MEDLINE/PubMed/PCM
- 化合物のデータベース: PubCem
今回はNCBIの主要なデータベースと解析ツールについて、いくつか基本の検索方法もあわせてご説明します。
(1) Nucleotide
Nucleotideには、GenBank(国際塩基配列データベース)、RefSeq(Reference Sequence:データ重複がなくアノテーションが付けられている二次的な核酸データベース)、TPA(Third Party Data:国際塩基配列データベースに登録されているエントリを元に第三者がアセンブルまたはアノテーションを行ったデータベース)などが含まれます。
実際にNucleotideデータベースを使用して、家族性アルツハイマー病の原因遺伝子として知られるAPP(Amyloid precursor protein)を例として遺伝子情報を検索してみましょう。
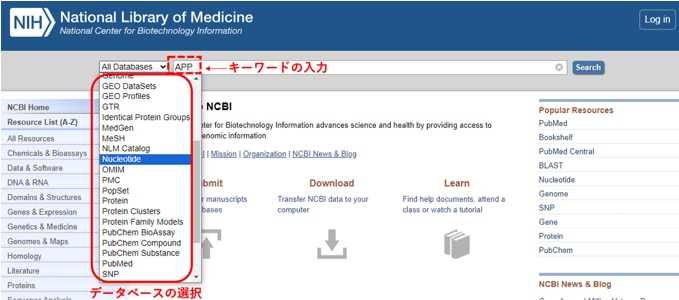
[図1. NCBIのTOP画面からの検索]
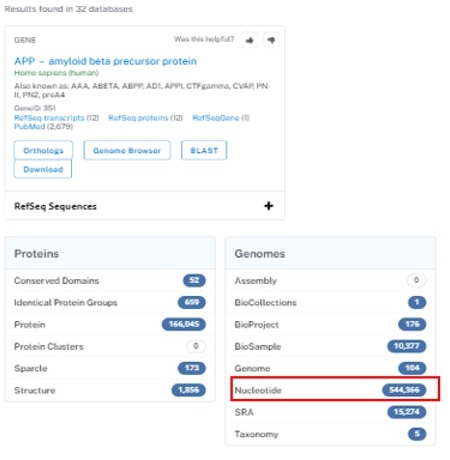
図1のようにNCBIのTOP画面の検索ボックスにキーワードを入力し、左にあるデータベースのリストをスクロールして”Nucleotide”のデータベースを選択してから検索すると、直接Nucleotideの検索結果が表示されます(図2上)。
またデフォルトの”All Database”で検索した場合、各種データベースの検索結果が表示されます(図3)。
表示された中より”Nucleotide”を選択すると先程と同様に核酸データベースにおける検索結果がでてきます(図2上)。
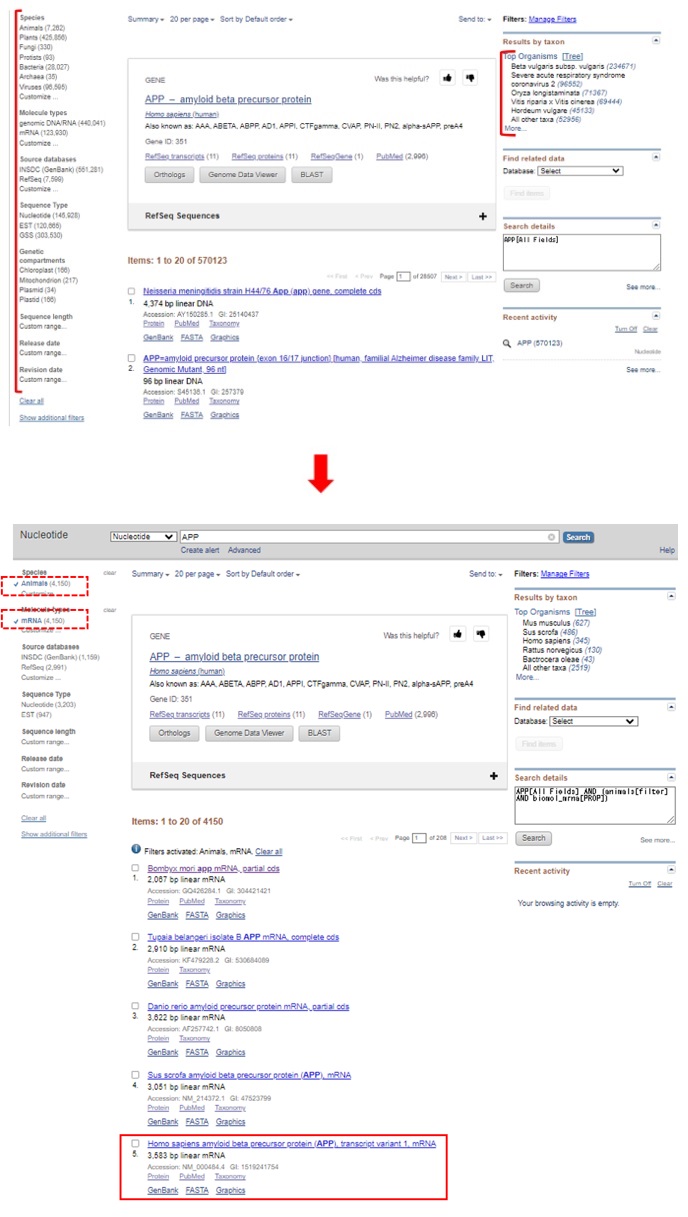
[図2. Nucleotideデータベースでの検索結果]
[図3. ”All Database”でキーワード検索した場合の検索結果]
検索結果の絞り込みを行うために、左端のフィルター機能より”Species”から”Animals”、”Molecule Type”から”mRNA”を指定、右端の”Result by Taxon”より”Homo sapiens ”と分類グループを指定することで、目的となる配列の絞り込みが行われます(図2下)。
また、検索式と検索フィールドを利用して検索ボックスに直接「APP AND Homo sapiens [Organism]」と入れても生物種の絞り込みができます。
次に、検索結果より5番目のタイトルを押下すると図4のデータが得られます。

[図4. 目的配列の検索結果]
図4のCDSはタンパク質のコード領域、ORIGINは塩基配列になります。
塩基配列については左上の”FASTA”よりFASTA形式で塩基配列データのファイルを取得することができます。
(2) Protein
GenBank、RefSeq、TPAのアノテーションされたコーディング領域を翻訳した配列情報、SwissProt、PIR(Protein Information Resource)、PRF(Protein Research Foundation)、PDB(Protein Data Bank)からの配列情報が含まれます。
Nucleotideと同様の方法で図2の”Protein”を選択してタンパク質情報が取得できます。
(3) dsSNP(single nucleotide polymorphism)
dbSNPはヒトの塩基多型に関するデータベースです。
一塩基変異、マイクロサテライト型(繰り返し配列の繰り返し回数による個人差)や挿入・欠失などの多型情報が含まれており、集団頻度の違いなど比較することができます。
実際にdsSNPを使用して、先ほど検索した「APP」を例としてSNP情報を検索してみましょう。
TOP画面よりキーワードを入力してSNPのデータベースを指定して検索すると、検索結果が表示されます。
“rs”ではじまるSNP IDやアレル、染色体位置、変異の種類、マイナーアレル頻度などの情報が確認できます(図5上)。
また、左側の項目をチェックすると検索の絞り込みができます。
候補となるSNP IDを押下すると様々な情報を確認できます。
”Variant Details”は基本情報、”Clinical Significance”ではSNPの臨床意義、”Frequency”では集団ごとのSNP頻度を確認することが可能です(図5下)。
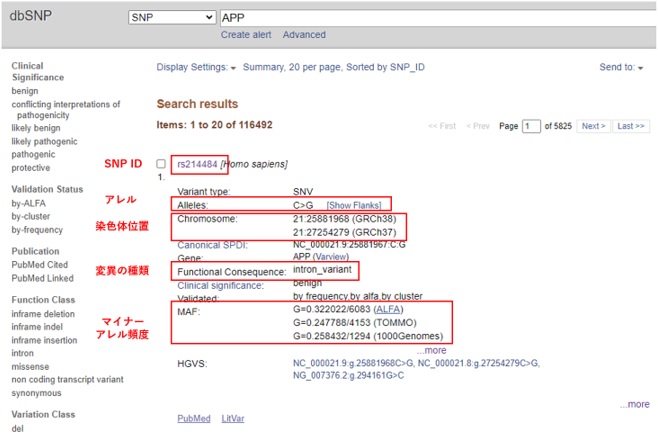
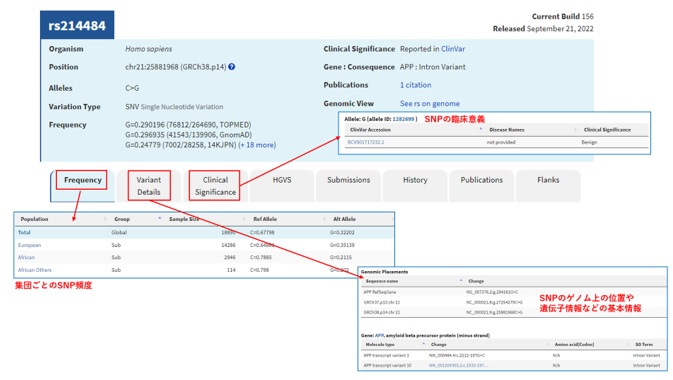
[図5. SNPの検索結果(上:APPのSNPの検索結果、下:個別のSNP情報詳細)]
3.解析ツール
主要な解析ツールとしてBLAST(Basic Local Alignment Search Tool)について、基本的な検索方法をご説明します。
(※BLASTのURL:https://blast.ncbi.nlm.nih.gov/Blast.cgi)
[図6. BLASTの種類]
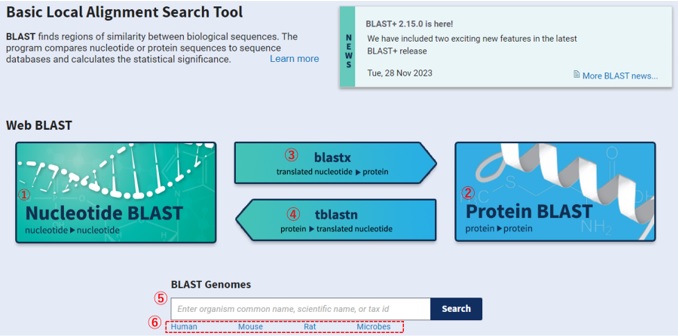
BLASTはNCBIが提供する配列データベースから問い合わせ配列(クエリ)に類似する配列を検索するツールで、以下の4種類のBLAST検索があります。
- Nucleotide BLAST(図6-①)
塩基配列を問い合わせ配列として、塩基配列データベースから検索 - Protein BLAST(図6-②)
アミノ酸配列を問い合わせ配列として、アミノ酸配列データベースから検索 - blastx(図6-③)
塩基配列を問い合わせ配列として、それをアミノ酸配列に翻訳したものをアミノ酸配列データベースから検索 - tblastn(図6-④)
アミノ酸配列を問い合わせ配列として、塩基配列データベースをアミノ酸に翻訳したものから検索
また、検索ボックスには生物種の一般名や学名を入力(図6-⑤)しますが、主要な生物種であるヒト、マウス、ラット、微生物(図6-⑥)については直接選択もできます。生物種を指定して配列類似性検索を行うことが可能です。
次に、“Nucleotido BLAST”を用いた検索方法を簡単に説明します。
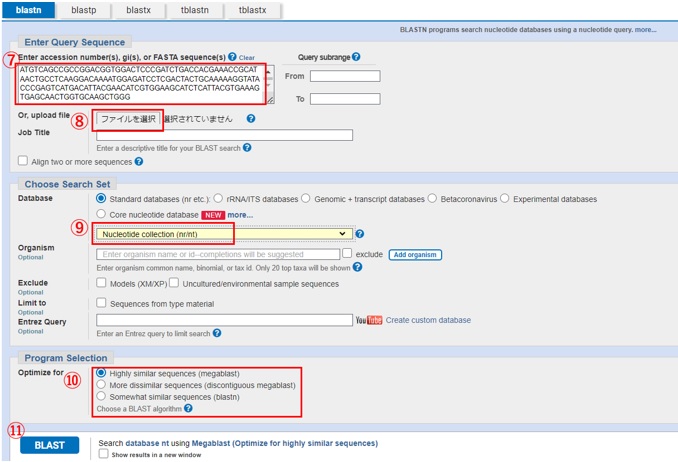
[図7. Nucleotide BLASTの検索方法]
まず、BLASTのTOP画面よりNucleotide BLAST(図6-①)を選択します。
検索画面でテキストボックス(図7-⑦)へ問い合わせの塩基配列を入力する、もしくはテキストファイルをファイル選択(図7-⑧)よりアップロードします。
検索対象のデータベースの種類(図7-⑨)と検索プログラム(図7-⑩)を選択して、” BLAST” (図7-⑪)を押下して検索すると類似配列がリストとなって表記されます(図8)。
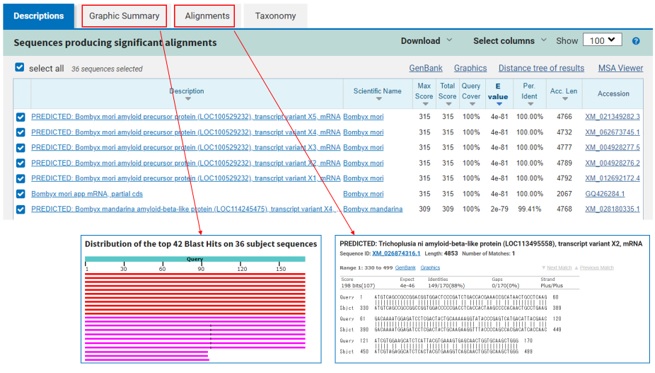
[図8: BLAST検索結果]
図8の ”Descriptions”では、アライメントスコアや一致率が確認でき、Accession#を押すと詳細を閲覧することが可能です。
”Graphic Summary”では視覚的に類似性を表示し、”Alignments”ではアライメント図が表示されます。
最後に“BLAST Patent Sequence”を用いて調べたい配列の類似配列を含むUS特許の有無について調べる方法を簡単に説明します。
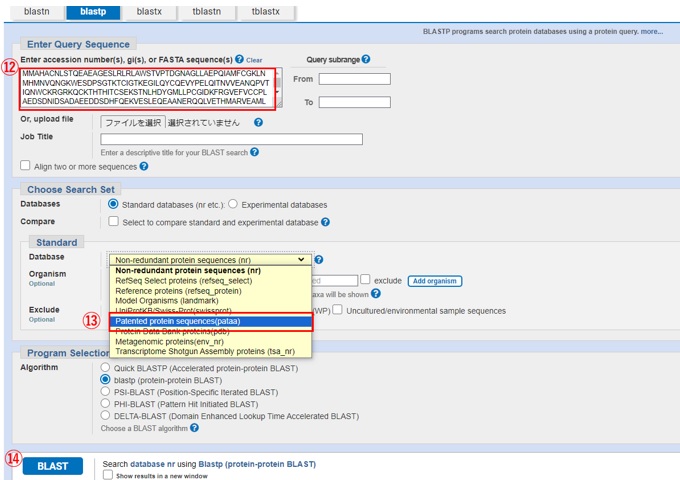
[図9: BLAST Patent Sequenceの検索方法]
BLASTのTOP画面よりProtein BLAST(図6-②)を選択します。
図7のNucleotido BLASTと同様に、検索画面でテキストボックス(図9-⑫)へ問い合わせの塩基配列を入力し、検索対象のデータベースの種類(図9-⑬)で「Patented Protein Sequences(pataa)」を選択して、” BLAST” (図9-⑭)を押下して検索すると類似配列の特許リストとなって表記されます(図10)。
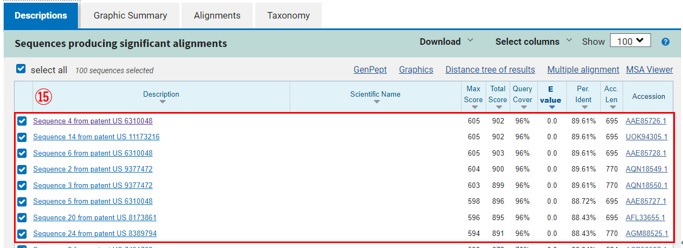
[図10: BLAST Patent Sequence検索結果]
ということで今回は、NCBIの代表的なデータベースとツールを使って簡単な検索を行ってみました。
取得した配列情報や周辺情報を利用して実際に実験ツールを作成することや、さらなる解析を行うことができます。
(日本アイアール株式会社 特許調査部 N・Y)















](https://engineer-education.com/wp/wp-content/uploads/2018/05/img_e-learning_tokkyo2_shingai-150x150.png)